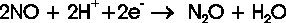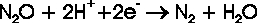|



|
Denitrifizierung
Der natürliche Kreislauf der Denitrifizierung umfaßt eine wichtige Kaskade von Enzymen, die stufenweise Nitrat
zu Distickstoff reduzieren [1]-[3]:

Die Denitrifizierung entspricht somit dem Teil des Stickstoffkreislaufs, welcher der Stickstoff-Fixierung
entgegengesetzt ist. Die Reduktion des Stickstoffmonoxids zu Distickstoffoxid erfolgt in der Natur durch
die NO-Reduktasen (NOR), die sowohl in Bakterien (NorBC) als auch in Pilzen (P450nor) vorkommen. Diese
Reaktion läuft nach folgender allgemeiner Reaktionsgleichung ab:
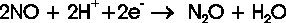
Die bakteriellen und fungalen Enzyme unterscheiden sich dabei fundamental in der Natur der aktiven Zentren,
und dementsprechend wurden für beiden Klassen ganz unterschiedliche Reaktionsmechanismen postuliert (siehe unten).
Im Kreislauf der Denitrifizierung wird Lachgas durch das Enzym N2O-Reduktase (N2OR) abgebaut,
wobei die Reaktion nach folgender allgemeiner Gleichung abläuft:
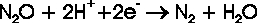
Im folgenden werden diese unterschiedlichen Klassen von Enzymen kurz vorgestellt.
Bakterielle NORs (NorBC)
Diese Enzyme sind eng mit den respiratorischen Häm-Kupfer Oxidasen, den sogenannten Cytochrom c-Oxidasen (CCO),
verwandt. Die bekannten Kristallstrukturen verschiedener CCOs können somit als Modell für die aktiven
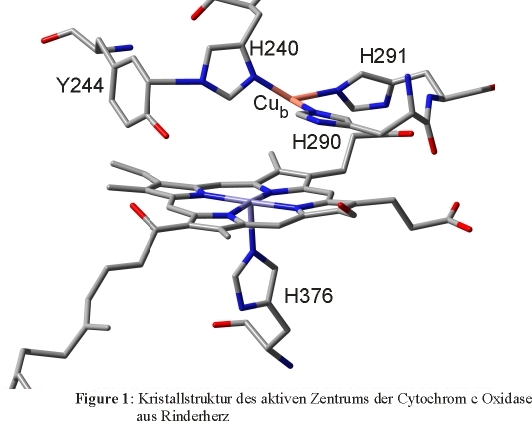
Zentren der bakteriellen NORs dienen. Abbildung 1 zeigt das aktive Zentrum von CCO aus Rinderherz [4], das aus einem
high-spin Häm a3 und dem sogenannten CuB-Zentrum besteht, das von drei Histidinen koordiniert ist.
Im Fall der NORs ist das aktive Zentrum strukturell sehr ähnlich und besteht im reduzierten Zustand aus einem fünffach
koordinierten high-spin Häm b (mit axialem Histidin) sowie einem Nicht-Häm Eisen, welches CuB im aktiven Zentrum
der CCOs ersetzt. Dieses wird daher auch als FeB bezeichnet.
Wie im Falle der CCOs ist über den molekularen Mechanismus der NORs bisher praktisch nichts bekannt. Eine Analyse
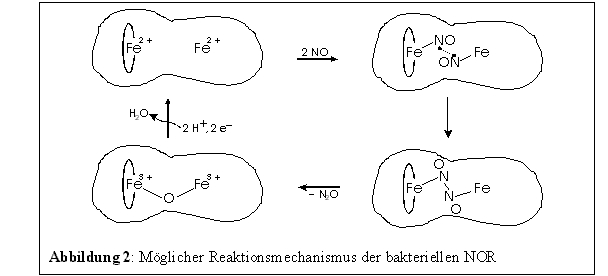
kinetischer Daten von NOR führte zu der Postulierung des Mechanismus' in Abbildung 2 [5]. Hierbei werden zunächst zwei
Moleküle NO gebunden und dann über die beiden Stickstoffatome gekoppelt. Nach der Abspaltung des Reaktionsprodukts
N2O sind die beiden nun oxidierten Eisenzentren über eine Oxid-Brücke verknüpft. Bisher konnte jedoch kein Intermediat
dieses Reaktionspfades charakterisiert werden, und dieser Mechanismus muß daher als rein spekulativ angesehen werden.
Alternativ könnte die Reduktion des NO auch ausschließlich am FeB ablaufen. Ein entsprechender Mechanismus wurde für
die Umsetzung von NO durch die CCOs postuliert, wo zwei Moleküle NO am CuB-Zentrum gebunden zu werden scheinen [6].
Umgekehrt scheint auch eine Reaktion nur an dem Häm b möglich zu sein, wie Untersuchungen an dem Modellsystem
[Fe(TPP)(NO)] (TPP = Tetraphenylporphyrin) ergeben haben [7].
Fungale NORs (P450nor)
Im Gegensatz zu den bakteriellen NORs, die eng mit den CCOs verwand sind, sind die fungalen NORs vom P450 abgeleitet
und werden daher auch als P450nor bezeichnet [8]. Die Kristallstruktur des Enzyms von Fusarium oxysporum zeigt ein Häm b
mit dem typischen axialen Cystein Thiolat-Liganden [9]. Verschiedene mechanistische Studien haben gezeigt, daß im Falle
von P450nor die Fe(III)-Form katalytisch aktiv ist. Bindung von NO führt zu einem sechsfach-koordinierten low-spin
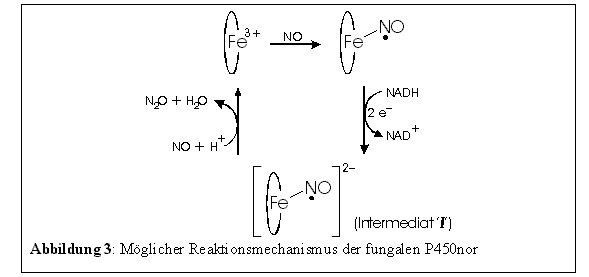
Komplex, der mit Hilfe verschiedener spektroskopischer Methoden charakterisiert wurde. Abbildung 3 zeigt einen möglichen
Mechanismus, der auf kinetischen Untersuchungen beruht [10]. Dabei wird der Fe(III)-NO Komplex zunächst mit zwei Elektronen
reduziert und die gebildete Spezies (Intermediat 'I') reagiert dann mit einem zweiten Molekül NO unter Bildung des Produkts
N2O.
N2OR
Die Kristallstruktur der N2O-Reduktase (N2OR) konnte für zwei unterschiedliche Organismen bestimmt werden.
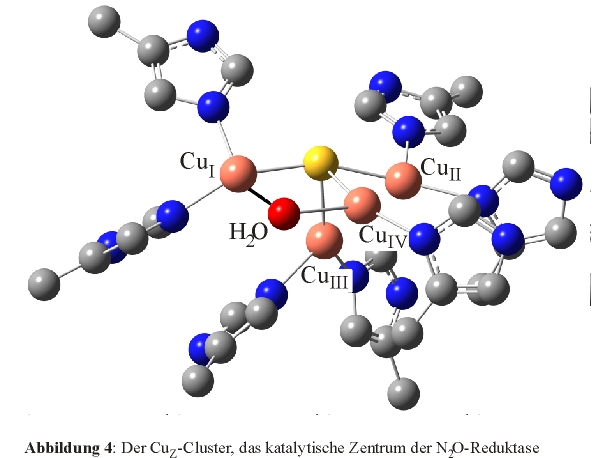
N2OR enthält einen einzigartigen Kupfer-Cluster, genannt CuZ [11], von dem man animmt, daß hier die Bindung und
Reduktion des N2O-Moleküls stattfindet. Wie in Abbildung 4 gezeigt, besteht der CuZ-Cluster
aus vier Kupfer-Zentren, die von einem Sulfid-Ion verbrückt werden, jedoch sind weder der Bindungsmodus des N2O
an das CuZ-Zentrum noch der Mechanismus der Reduktion zu N2 bekannt [12],[13].
Literatur:
[1] Ferguson, S. J. Curr. Opin. Chem. Biol. 1998, 2, 182-193.
[2] Richardson, D. J.; Watmough, N. J. Curr. Opin. Chem. Biol. 1999, 3, 207-219.
[3] Moura, I.; Moura, J. J. G. Curr. Opin. Chem. Biol. 2001, 5, 168-175.
[4] Tsukihara, T.; Aoyama, H.; Yamashita, E.; Tomizaki, T.; Yamaguchi, H.; Shinzawa-Itoh, K.; Nakashima, R.; Yaono, R.; Yoshikawa, S.
Science 1996, 272, 1136-1144.
[5] Zumft, W. J. Inorg. Biochem. 2005, 99, 194-215.
[6] Butler, C. S.; Seward, H. E.; Greenwood, C.; Thomson, A. J. Biochemistry 1997, 36, 16259-16266.
[7] Lin, R.; Farmer, P. J. J. Am. Chem. Soc. 2001, 123, 1143-1150.
[8] Daiber, A.; Shoun, H.; Ullrich, V. J. Inorg. Biochem. 2005, 99, 185-193.
[9] Park, S.-Y.; Shimizu, H.; Adachi, S.-I.; Nakagawa, A.; Tanaka, I.; Nakahara, K.; Shoun, H.; Obayashi, E.; Nakamura, H.; Iizuka, T.; Shiro, Y.
Nature Struct. Biol. 1997, 4, 827-832.
[10] Shiro, Y.; Fujii, M.; Iizuka, T.; Adachi, S.-I.; Tsukamoto, K.; Nakahara, K.; Shoun, H.
J. Biol. Chem. 1995, 270, 1617-1623.
[11] ) Haltia, T.; Brown, K.; Tegoni, M.; Cambillau, C.; Saraste, M.; Mattila, K.; Djinovic-Carugo, K.
Biochem. J. 2003, 369, 77-88.
[12] Paulat, F.; Kuschel, T.; Näther, C.; Praneeth, V. K. K.; Sander, O.; Lehnert, N. Inorg. Chem. 2004, 43, 6979-6994.
[13] Chen, P.; Gorelsky, S. I.; Ghosh, S.; Solomon, E. I. Angew. Chem. Int. Ed. 2004, 43, 4132-4140.
|